
덴하덴하 ~♬ 덴싸입니다.
이번에 다룰 것은 matching sampling 하는 방법인데요 그 중에서도
propensity score matching 방법을 이용해서 샘플링을 해볼거예유~
우선 샘플링에 필요한 데이터를 불러보쥬
mydata <- read.csv("D:/google/workground/datasea/2019cha/tbl_IV.csv", fileEncoding = "utf-8", stringsAsFactors = F)
str(mydata)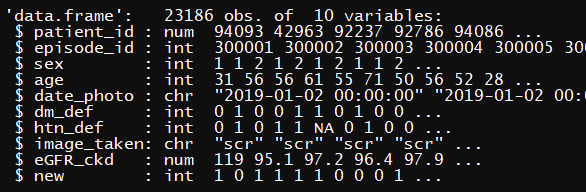
대충 이런 구조를 가지고 있어유 나같은 경우는 new ==1 이면서 eGFR_ckd 60 미만 그룹과 그렇지 않은 그룹으로 샘플링을 할거에유
일단 mutate function을 이용해서 abnormal로 조건 그룹 체크를 해주고 매칭에 필요한 변수("age", "sex", "abnormal")의 missing value를 제거 해줄게유
mydata_mu <- mydata %>%
mutate(abnormal = ifelse(eGFR_ckd < 60 & new == 1 , T, F)) %>%
filter(new == 1) %>%
filter(!is.na(abnormal)) %>%
select(patient_id,episode_id,sex,age,abnormal,image_taken) %>%
mutate(image_taken = as.factor(image_taken))
str(mydata_mu)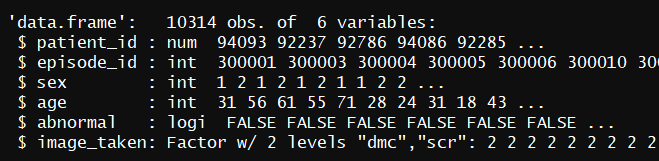
조건으로 그룹이 나눴는디 두 집단이 현재 어떤 차이를 보이는지 카이제곱 테스트를 확인해 볼거에유
install.packages("pacman")
pacman::p_load(tableone)
t1 <- CreateTableOne(vars = c("age", "sex"),
data = mydata_mu,
factorVars = "sex",
strata = "abnormal") %>%
print()
kable(t1[,1:3], align = 'c', caption = "age, sex")
p value가 0.05 보다 작으니 두 sample은 unmatched sample이라 말할 수 있어유
자 그럼 matching sampling에 필요한 MatchIt function을 이용해서 mathcing sampling을 해보쥬
install.packages("MatchIt")
library(MatchIt)
set.seed(200615)
mi <- matchit(abnormal ~ age + sex, data = mydata_mu, method = "nearest", ratio = 1)
summary(mi)
plot(mi, type = "jitter", interactive = F)"MatchIt" 패키지 깔고 library에 올려주시고 set.seed(숫자)로 씨드넘버 설정해유
아까 그룹화 변수로 이용한 abnormal을 먼저 적어주고 group variable은 반드시 logic variable이여야 해유
그리고 매칭할 변수들을 적어유(age + sex)
method 는 nearest neighbors 방법 말고도 c("exact", "subclass", "optimal", ""genetic", "full")이 존재해유
ratio는 group variable 상 TRUE로 잡혀있는 수를 기준으로 FALSE 그룹의 비율을 뜻해유
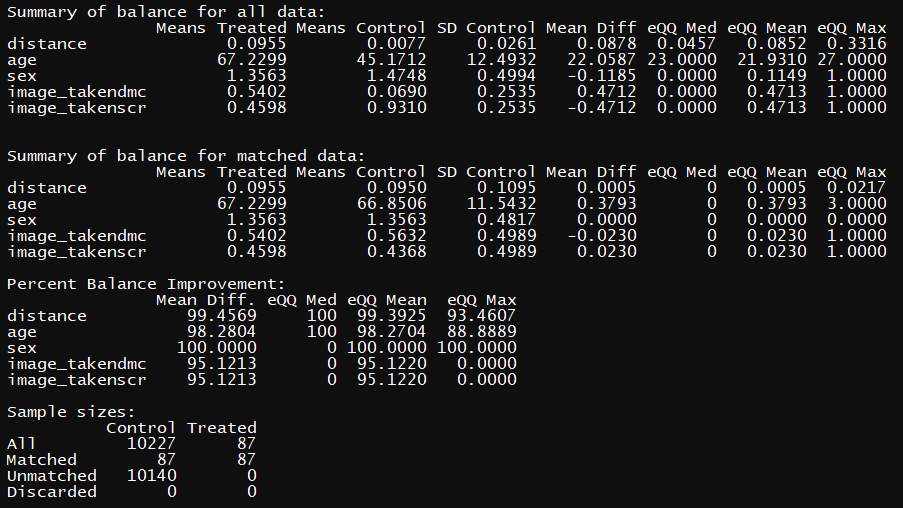
그럼 이런 내용들로 matching 작업을 했다고 서머리가 나와유
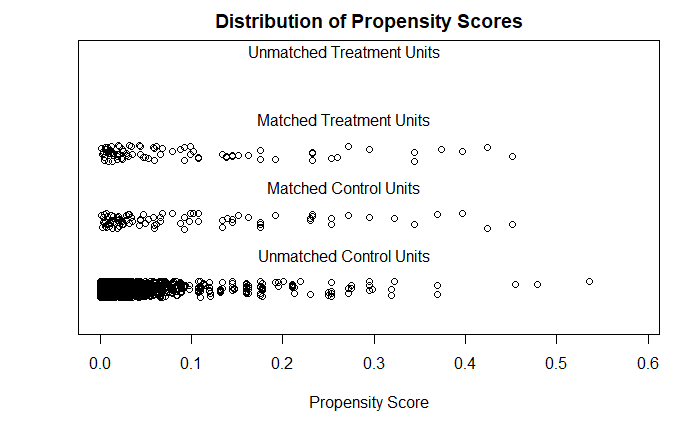
plot으로도 score 분포를 어떤지 한번에 확인가능해유
그럼 매칭된 샘플들을 저장해 보아유 match.data()를 이용해유
매칭된 sample 데이터에 해당하는 episode_id를 뽑아와서 저장 시켜유~
sample_matched <- match.data(mi)
df_sample <- mydata %>%
filter(episode_id %in% sample_matched$episode_id) %>%
select(-"new")
write.csv(df_sample, "D:/google/workground/datasea/2019cha/tbl_IV_matched_v1.csv")
제대로 matching이 됐는지 확인해봐야겠쥬?
t4 <- CreateTableOne(vars = c("age","sex"),
data = sample_matched,
factorVars = "sex",
strata = "abnormal") %>%
print()
kable(t4[,(1:3)], align = "c")
확인해보면 p-value 상 두 집단의 age와 sex의 distribution은 같다고 판단할 수 있네유~
